Clustering et classification
- Julien Duval
- R , Data
- 5 avril 2022
Sommaire
Application de méthodes de clustering et de classification à divers ensembles de données
Travail sur le clustering et la classification
Ce travail pratique porte sur l’application de méthodes de regroupement et de classification à divers ensembles de données.
Clustering et Classification sur le dataset Iris
- Apply clustering methods to the Iris dataset.
- Visualize and interpret the results.
- Use the k-NN method to create a confusion matrix.
library(class)
library(ggplot2)
library(dbscan)
# Chargement des données iris
data(iris)
# Suppression de la colonne Species pour appliquer les algorithmes
iris_data <- iris[, -5]
iris_labels <- iris$Species
#----------------------------K-Means--------------------------------------------
#### Appliquer k-means sur les données iris
set.seed(42)
kmeans_result <- kmeans(iris_data, centers = 3)
# Ajouter les clusters prévus dans les données
iris$Cluster_kmeans <- as.factor(kmeans_result$cluster)
#------------------------hierarchical clustering--------------------------------
dist_matrix <- dist(iris_data) # Matrice de distances
hclust_result <- hclust(dist_matrix, method = "ward.D2")
# Couper l'arbre pour obtenir 3 clusters
hclust_clusters <- cutree(hclust_result, k = 3)
iris$Cluster_hclust <- as.factor(hclust_clusters)
#-----------------------------DBSCAN--------------------------------------------
# eps = rayon maximal d'un voisinage) et minPts (nombre minimum de voisins)
dbscan_result <- dbscan(iris_data, eps = 0.5, minPts = 5)
# Ajouter les clusters DBSCAN à la dataframe iris
iris$Cluster_dbscan <- as.factor(dbscan_result$cluster)
#-------------------Séparation des données en train et test---------------------
# Séparation des données en train et test
set.seed(42)
train_idx <- sample(1:nrow(iris_data), 0.7 * nrow(iris_data))
train_data <- iris_data[train_idx, ]
train_labels <- iris_labels[train_idx]
test_data <- iris_data[-train_idx, ]
test_labels <- iris_labels[-train_idx]
# Appliquer la méthode K-NN avec k = 3
knn_pred <- knn(train = train_data, test = test_data, cl = train_labels, k = 3)
#-------------------Visualisation des résultats---------------------------------
##K-means
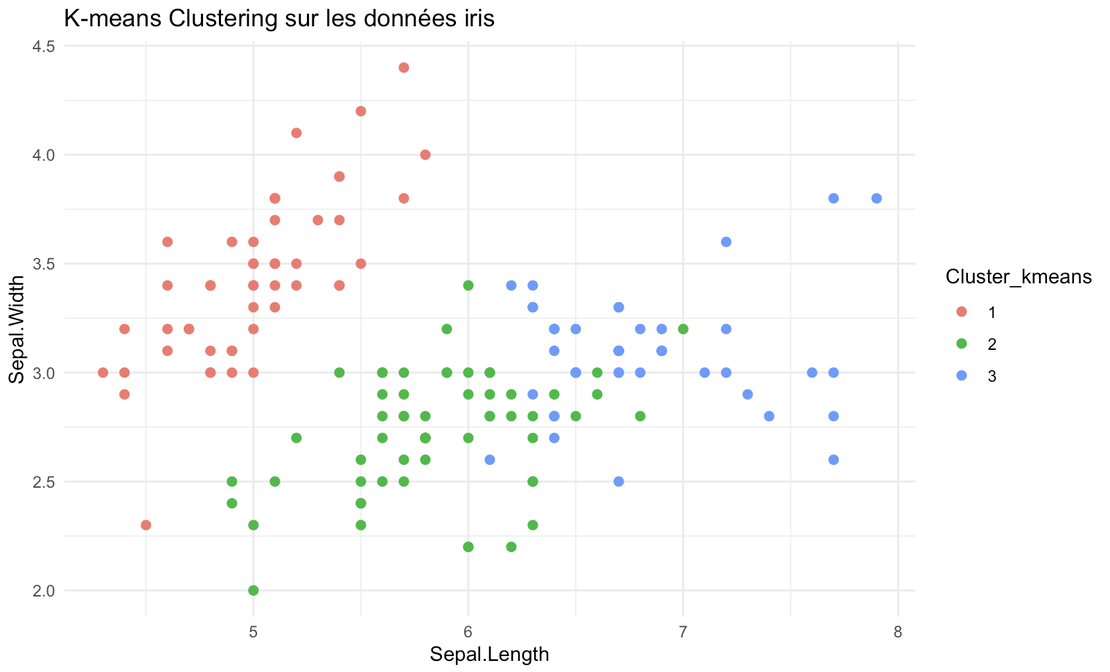
ggplot(iris, aes(x = Sepal.Length, y = Sepal.Width, color = Cluster_kmeans)) +
geom_point(size = 2) +
labs(title = "K-means Clustering sur les données iris") +
theme_minimal()
##Hierarchical Clustering
ggplot(iris, aes(x = Sepal.Length, y = Sepal.Width, color = Cluster_hclust)) +
geom_point(size = 2) +
labs(title = "Hierarchical Clustering sur les données iris") +
theme_minimal()
##DBSCAN
ggplot(iris, aes(x = Sepal.Length, y = Sepal.Width, color = Cluster_dbscan)) +
geom_point(size = 2) +
labs(title = "DBSCAN Clustering sur les données iris") +
theme_minimal()
##K-NN - sur les données de test uniquement
test_results <- data.frame(Sepal.Length = test_data$Sepal.Length,
Sepal.Width = test_data$Sepal.Width,
Predicted = knn_pred)
ggplot(test_results, aes(x = Sepal.Length, y = Sepal.Width, color = Predicted)) +
geom_point(size = 2) +
labs(title = "K-NN method sur les données iris (test)") +
theme_minimal()
#-----------------------Matrice de confusion------------------------------------
# Matrice de confusion pour K-NN
table(Predicted = knn_pred, Actual = test_labels)
# Matrice de confusion pour K-means
table(Kmeans = kmeans_result$cluster, Réel = iris_labels)
# Matrice de confusion pour DBSCAN
table(DBSCAN = dbscan_result$cluster, Réel = iris_labels)
# Matrice de confusion pour hclust
table(Hclust = hclust_clusters, Réel = iris_labels)
Analyse du dataset Diabetes
- Examine the diabetes dataset.
- Apply clustering methods to retrieve the initial classification.
- Analyze the quality of results using raw and standardized data.
- Attempt classification using k-NN and comment on the results.
library(class)
library(ggplot2)
library(dbscan)
# Charger les données
data <- read.csv("/Machine Learning/Clustering/dataset_diabetes.csv",
header = TRUE, sep = ",")
# Sélectionner les variables numériques pour le clustering
X_data <- data[, sapply(data, is.numeric)]
# Données brutes
X_raw_data <- X_data
# Données centrées et scalées
X_scaled_data <- scale(X_data)
# Assurez-vous que la colonne cible existe
y_data <- data$class # Ajustez "class" au bon nom de colonne
#----------------------------K-Means--------------------------------------------
# K-Means sur les données brutes
set.seed(100)
kmeans_result_raw <- kmeans(X_raw_data, centers = 2)
data$Cluster_kmeans_raw <- as.factor(kmeans_result_raw$cluster)
# K-Means sur les données normalisées
kmeans_result_scaled <- kmeans(X_scaled_data, centers = 2)
data$Cluster_kmeans_scaled <- as.factor(kmeans_result_scaled$cluster)
#------------------------Hierarchical Clustering--------------------------------
# Hierarchical Clustering sur les données brutes
dist_matrix_raw <- dist(X_raw_data) # matrice de distances pour les données brutes
hclust_result_raw <- hclust(dist_matrix_raw, method = "ward.D2")
hclust_clusters_raw <- cutree(hclust_result_raw, k = 2)
data$Cluster_hclust_raw <- as.factor(hclust_clusters_raw)
# Hierarchical Clustering sur les données normalisées
dist_matrix_scaled <- dist(X_scaled_data) # matrice de distances pour les données scalées
hclust_result_scaled <- hclust(dist_matrix_scaled, method = "ward.D2")
hclust_clusters_scaled <- cutree(hclust_result_scaled, k = 2)
data$Cluster_hclust_scaled <- as.factor(hclust_clusters_scaled)
#-----------------------------DBSCAN--------------------------------------------
# DBSCAN sur les données scalées et centrées
dbscan_result_scaled <- dbscan(X_scaled_data, eps = 2.5, minPts = 15)
data$Cluster_dbscan_scaled <- as.factor(dbscan_result_scaled$cluster)
# Visualisation de l'eps avec KNNdistplot pour les données normalisées
kNNdistplot(X_scaled_data, k = 8)
abline(h = 2, col = "red")
#-------------------Séparation des données en train et test---------------------
# Séparer les données en train et test (sur les données scalées et centrées)
set.seed(100)
train_idx <- sample(1:nrow(X_scaled_data), 0.7 * nrow(X_scaled_data))
train_data <- X_scaled_data[train_idx, ]
train_labels <- y_data[train_idx]
test_data <- X_scaled_data[-train_idx, ]
test_labels <- y_data[-train_idx]
# Appliquer la méthode K-NN avec k = 2
knn_pred <- knn(train = train_data, test = test_data, cl = train_labels, k = 2)
#-------------------Visualisation des résultats---------------------------------
## K-Means sur les données brutes
ggplot(data, aes(x = pedigree, y = glucose, color = Cluster_kmeans_raw)) +
geom_point(size = 2) +
labs(title = "K-means Clustering sur les données brutes") +
theme_minimal()
## K-Means sur les données normalisées
ggplot(data, aes(x = pedigree, y = glucose, color = Cluster_kmeans_scaled)) +
geom_point(size = 2) +
labs(title = "K-means Clustering sur les données normalisées") +
theme_minimal()
## Hierarchical Clustering sur les données brutes
ggplot(data, aes(x = pedigree, y = glucose, color = Cluster_hclust_raw)) +
geom_point(size = 2) +
labs(title = "Hierarchical Clustering sur les données brutes") +
theme_minimal()
## Hierarchical Clustering sur les données normalisées
ggplot(data, aes(x = pedigree, y = glucose, color = Cluster_hclust_scaled)) +
geom_point(size = 2) +
labs(title = "Hierarchical Clustering sur les données normalisées") +
theme_minimal()
## DBSCAN sur les données normalisées
ggplot(data, aes(x = pedigree, y = glucose, color = Cluster_dbscan_scaled)) +
geom_point(size = 2) +
labs(title = "DBSCAN Clustering sur les données normalisées") +
theme_minimal()
## K-NN sur les données de test
test_data <- as.data.frame(test_data)
test_results <- data.frame(pedigree = test_data$pedigree,
glucose = test_data$glucose,
Predicted = knn_pred)
ggplot(test_results, aes(x = pedigree, y = glucose, color = Predicted)) +
geom_point(size = 2) +
labs(title = "K-NN method sur les données (test)") +
theme_minimal()
#-----------------------Matrices de Confusion-----------------------------------
# Matrice de confusion pour K-NN
table(Predicted = knn_pred, Actual = test_labels)
# Matrice de confusion pour K-means sur les données brutes (comparaison qualitative)
table(Kmeans_raw = data$Cluster_kmeans_raw, Réel = y_data)
# Matrice de confusion pour K-means sur les données normalisées (comparaison qualitative)
table(Kmeans_scaled = data$Cluster_kmeans_scaled, Réel = y_data)
# Matrice de confusion pour DBSCAN (comparaison qualitative)
table(DBSCAN_scaled = data$Cluster_dbscan_scaled, Réel = y_data)
# Matrice de confusion pour hclust sur les données brutes (comparaison qualitative)
table(Hclust_raw = data$Cluster_hclust_raw, Réel = y_data)
# Matrice de confusion pour hclust sur les données normalisées (comparaison qualitative)
table(Hclust_scaled = data$Cluster_hclust_scaled, Réel = y_data)
